haje01 노트
스네이크메이크 (Snakemake) 튜토리얼
목차
워크플로우 관리가 필요한 이유빠르게 시작하기
파일내 단어 수 세기
개별 단어 수 파일 병합
그래프 그리기
기본 규칙 만들기
DAG 시각화하기
DAG 동작 돌아보기
심화
두 가지 의존성
데이터 의존성
워크플로우 의존성
사전형으로 입출력 기술하기
규칙에 매개변수 이용하기
코드에서 와일드카드 참조하기
특정 디렉토리내 모든 파일을 입력으로 하기
와일드카드에 정규식으로 제약걸기
S3 에서 파일 입출력
Jupyter 노트북 실행하기
외부 워크플로우 파일 이용하기
설정 파일 이용하기
스크립트 파일 합치기?
기타 팁들
쉘 명령어 여러 줄 사용하기
더미 출력 파일 이용하기
강제로 모든 규칙 실행하기
고정된 출력 파일의 입력을 함수에서 얻기
체크포인트 이용하기
빌드 결과물 모두 지우기
메모리 문제 대응
마무리
스네이크메이크 (Snakemake) 튜토리얼
데이터 과학이나 엔지니어링을 하다보면 점점 더 복잡해지는 데이터와 코드의 숲에서 헤메는 경우가 생긴다. 이럴 때는 데이터를 체계적으로 처리하게 해주는 워크플로우 관리 시스템을 도입하는 것이 좋은 해결책이 될 수 있다.
이 글에서는 파이썬용 워크플로우 관리 시스템들 중 하나인 Snakemake (스네이크메이크) 에 대해 실습을 통해 알아보겠다. Snakemake 버전 5.14 와 유닉스 계열 (Linux/macOS) OS 관점에서 설명하겠지만, 경로 구분자 등 몇몇 차이를 고려하면 윈도우에서도 적용에 무리가 없을 것이다.
워크플로우 관리가 필요한 이유
요즘은 데이터 과학과 인공지능이 각광받는 시대이지만, 사실 그것을 위한 준비 과정의 어려움은 간과되기 쉽다. 그중 하나가 데이터 엔지니어링인데, 간단한 데이터 처리라면 그다지 복잡할 것이 없겠지만, 현업에서 다양한 소스의 데이터를 처리 (ETL) 하고 고도화된 피처 엔지니어링을 수행하다 보면 체계적인 작업 관리의 필요성을 느낄 수 있다. 대표적인 문제 몇 가지를 나열하면:
- 다양한 데이터 소스 간 의존성 파악이 힘듦 - 작업에 필요한 데이터가 모두 준비되어 있는지 확인이 번거로움
- 반복적인 데이터 처리작업으로 인한 비효율성 - 꼭 필요하지 않은 전처리를 반복하는 경우
- 처리 코드와 의존 관계 혼재 - 데이터 간 의존성 관리 및 처리 코드가 혼재되어 있어 재활용성이 낮아지고 가독성이 떨어짐
이와 같은 문제를 해결하기 위해 데이터 처리를 위한 워크플로우 관리 시스템 (Workflow Management System, WMS) 이 활용되고 있다. 대부분의 워크플로우 매니저는 유향 비순환 그래프 (Directed Acyclic Graph, DAG) 방식으로 구현되어 있다. 아래에 간단한 DAG 구조의 예를 그림로 나타내었다.
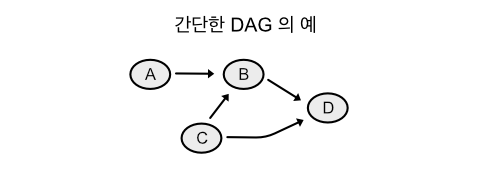
DAG 에서 각 노드는 작업을 의미하고, 자기 작업을 수행하기 위해 필요한 의존 작업의 노드에 연결되어 있다. 위 그림에서 최종 타겟 D를 수행하기 위해, B 와 C 가 필요하고, B 가 수행되기 위해서는 A 와 C 가 수행되어야 한다. 타겟 결과물이 요청되면 그것을 생성하기 위해 필요한 의존 작업을 재귀적으로 거슬러 올라가 수행하는 방식인데, 위의 경우 A, C 가 먼저 수행되고 다음이 B, 끝으로 D 가 수행된다. 대부분 WMS 가 이 DAG 를 통해 의존관계를 해결해 나가는 방식으로 구현되어 있다.
요즘은 AirFlow, Luigi, Snakemake 등 다양한 WMS 가 쓰이고 있는데, 이 글에서는 Snakemake (스네이크메이크) 에 대해서 알아본다. 필자가 생각하는 Snakemake 의 장점은 아래와 같다.
- GNU Make 형식의 컴팩트한 구문
- 데이터 의존관계 파악이 쉽다.
- 처리 코드와 의존 관계 명세를 나누어 재활용성이 높다.
- 파이썬으로 구현되어 호환성이 좋다.
요즘처럼 통합개발환경 (IDE) 이 없었던 옛날에는, 프로그램을 빌드하기 위해서 여러 가지 소스 코드 파일과 중간 산출물 파일들을 직접 관리해 주어야 했는데 그것은 꽤 복잡한 과정이었다. 이를 해결하기 위해서 사용된 것이 GNU Make 인데, Snakemake는 그것의 철학을 이어받은 WMS 라고 할 수 있겠다.
의존성을 해결하며 최종 타겟을 만들어 가는 과정을 Make 의 관례에 따라 빌드 라고 하겠다.
Snakemake 는 이름이 Snakefile 인 워크플로우 파일 에 데이터의 의존 관계와 실행 정보를 코드와 별도로 기술한다. 의존 관계와 처리 코드를 나누는 것에 대해 설명하기 위해, 아래에 DAG 를 데이터 의존 관계와 처리 코드, 그리고 생성된 데이터로 구분해 그려보았다.
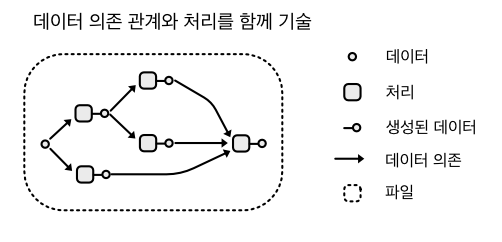
그림에서 네모는 처리 코드이고, 속이 빈 점은 데이터, 그리고 화살표는 데이터 의존관계를 나타낸다. 둘러싼 점선은 파일로 생각하면 되는데, 위의 경우 데이터 의존성과 처리가 하나의 파일에 함께 기술되었다. 일반적인 파이썬 코드로 데이터 처리를 구현하면 위와 같은 모습이 될 것이다. Snakemake 를 활용해 두 가지를 분리하면 아래와 같이 된다.
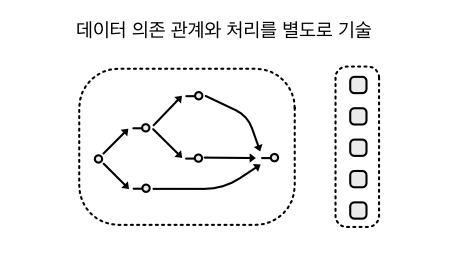
데이터 관계와 처리 코드가 나누어져 보다 명료해진 것이 느껴진다. 이렇게 하면 데이터 의존 관계를 파악하기 쉽고, 코드는 재활용성이 높아지는 효과를 기대할 수 있다.
실제로 잘 문서화된 워크플로우 파일은 데이터 목록 (Data Catalog) 역할을 할 수도 있을 것이다.
이제 간단한 튜토리얼을 통해 Snakemake 에 대해 알아보자. 여담이지만, Snakemake 의 가장 좋은 정보원은 Snakemake 공식 문서 라고 생각한다. 그런데 아쉬운 점은 Snakemake의 개발자가 Bio Informatics 를 전문으로 하고 있어서, 문서의 예제가 바이오 쪽의 툴과 데이터를 이용하고 있다는 점이다. 아무래도 그 분야에 익숙하지 않은 사람들에게는 생소하기 때문에, 이 글에서는 가급적 일반적인 예로 설명하려 한다.
빠르게 시작하기
먼저 Snakemake 의 설치가 필요한데, 일반적으로 다음처럼 pip 를 이용하여 간단히 설치 가능하다.
$ pip install snakemake
그렇지만 현재 (2024-02-16 기준) 최신 Snakemake 인 버전 8.x 는 상당히 많은 부분이 바뀌고 아직 성숙되지 않은 듯 하여, 이 글에서는 다음과 같이 설치된 Snakemake 를 기준으로 하여 설명하겠다.
pip install PuLP==2.3.0
pip install snakemake==7.15.2
PuLP는 Snakemake 에서 사용하는 외부 패키지인데 최신 버전에 버그가 있어, 버그가 없는 버전으로 먼저 설치한 후 Snakemake 를 설치하는 과정이다.
예제를 위해 아래의 패키지도 설치하자.
pip install pandas==1.5.3
pip install matplotlib==3.7.4
앞으로 설명할 예제는 각 파일에 들어있는 단어의 수를 세고, 최종적으로 파일별 단어 수 그래프를 만드는 것을 목표로 한다.
먼저 데이터 디렉토리가 다음과 같이 구성되어 있다고 하자:
data/
A.txt
B.txt
C.txt
파일의 내용은 아래와 같다.
A.txt
Hello.
B.txt
This is your captain speaking.
C.txt
Welcome aboard.
이를 처리하기 위해 먼저 워크플로우 파일인 Snakefile 이 필요하다. Snakemake 는 이 워크플로우 파일의 내용대로 동작하게 된다.
앞에서 설명한 것처럼 워크플로우는 하나 이상의 작업 노드들로 구성되어 있는데, Snakemake 에서는 이 노드를 규칙 (Rule) 이라 부르며, Snakefile 에서 rule 이라는 키워드로 선언한다. 규칙은 기본적으로 다음과 같은 구조를 가진다.
rule 규칙 이름:
입력 파트:
출력 파트:
실행 파트:
파트별 구체적인 키워드는 다음과 같다.
rule RULE_NAME:
input:
output:
run, script, shell, notebook 중 선택:
rule에는 규칙의 이름이 온다.input에는 규칙의 입력이 되는 하나 이상의 파일 경로나, 코드 또는 함수가 온다. 경로가 하나 이상인 경우,를 사용하여 구분한다.output에는 규칙의 출력이 되는 하나 이상의 파일 경로가 온다. 하나 이상인 경우,를 사용하여 구분한다.- 실행 파트에는 타입별로 다음과 같은 내용이 온다.
shell- 쉘 명령을 기술run- 파이썬 스크립트를 직접 기술script- 파이썬 스크립트 파일 경로를 지정notebook- Jupyter 노트북 파일 경로를 지정
Snakefile은 기본적으로 파이썬 파일과 같다. 즉, 파이썬 모듈을 임포트하거나 함수를 정의하는 등 파이썬 명령어를 사용할 수 있다. 단, 위에서 보듯 규칙 부분은 다른 구문을 사용한다.
파일내 단어 수 세기
이제 다음과 같은 내용으로 Snakefile 을 현재 디렉토리에 만들어 보자:
rule count:
"""파일내 단어 수 세기."""
input:
"data/{filename}.txt"
output:
"temp/wc_{filename}.txt"
shell:
"wc -w {input} > {output}"
위 예는 count 라는 규칙을 정의한다. data/ 디렉토리 아래의 [파일명].txt 에 매치되는 모든 파일을 입력으로 하여 shell 에 기술된 쉘 명령을 수행한 후, 그 결과를 temp/wc_[파일명].txt 파일에 출력한다는 뜻이다.
wc -w는 파일에서 단어를 세는 Unix 계열 쉘 명령어다. 예를 들면 다음과 같다.$ wc -w data/B.txt 5 data/B.txt
B.txt에 다섯 단어가 있음을 알 수 있다.
입력 및 출력에서 {filename} 부분은 와일드카드 (Wildcard) 로 불리는데, 패턴이 매칭되는 모든 파일 경로에서 대체되는 변수이다.
Snakemake 의 실행은 Snakefile 이 있는 디렉토리에서 다음과 같이 한다.
$ snakemake [빌드 타겟] -j [숫자]
빌드 타겟은 Snakemake 를 실행하여 얻고자 하는 타겟 파일을 지정한다.
-j 옵션은 --jobs 또는 --cores 의 단축형으로 숫자 인자를 받는다. 이것은 Snakemake 빌드에서 최대 몇 개의 CPU 코어를 활용할 것인지 명시하는 것이다. 복수의 코어를 사용하면 DAG 에 따라 의존성이 없는 규칙들은 병렬로 처리하여 속도를 올릴 수 있다. 숫자를 생략하면 가용한 최대 코어를 이용한다.
예제를 위해 다음과 같이 실행해보자:
$ snakemake temp/wc_A.txt -j
위 명령은 temp/wc_A.txt 를 타겟으로 빌드하라는 것인데, 이를 위해서는 출력 ( output ) 이 temp/wc_A.txt 인 규칙이 필요하다. 여기서 그 규칙은 count 이다. 규칙이 매칭되면 와일드카드 {filename} 은 A 로 배정되며, 이에 의해 입력 ( input ) 은 data/A.txt 로 결정된다. 결과적으로 실행은 쉘명령어 wc -w data/A.txt > temp/wc_A.txt 를 수행하게 된다. 실행 후 다음처럼 결과를 확인해보자:
$ cat temp/wc_A.txt
1 data/A.txt
data/A.txt 파일의 단어 수가 잘 저장되었다.
이처럼 Snakemake는 각 규칙의 결과물을 파일로 저장하는데, 이를 통해 반복 수행을 피하고 중간 결과 확인이 용이해진다.
규칙의 출력을 입력으로 이용할 다른 규칙이 있으면 자동으로 빌드가 진행되기에, 모든 타겟을 명시적으로 빌드해줄 필요는 없다.
개별 단어 수 파일 병합
다음 단계는 파일별 단어 수를 하나의 .csv 파일에 저장하는 것이다. 이를 위해 Snakefile 에 concat 규칙을 추가한다.
rule concat:
"""개별 단어 수 파일을 병합."""
input:
"temp/wc_A.txt",
"temp/wc_B.txt",
"temp/wc_C.txt"
output:
"temp/wc_all.csv"
script:
"concat.py"
위 규칙은 앞에서 생성한 모든 단어 수 파일을 입력으로 한다. 즉, count 규칙은 입력과 출력 파일이 일대일 의 관계였지만 concat 규칙은 다대일 의 관계이다.
input 에 모든 단어 수 파일을 명시하고 있는데, 이렇게 해도 되지만 다음처럼 expand 함수를 사용하면 편리하다.
rule concat:
"""개별 단어 수 파일을 병합."""
input:
expand("temp/wc_{filename}.txt", filename=['A', 'B', 'C'])
output:
"temp/wc_all.csv"
script:
"concat.py"
expand 는 매개변수로 받은 리스트의 각 항목으로 패턴을 확장한다. 위의 경우, 결과는 아래와 같다.
["temp/wc_A.txt", "temp/wc_B.txt", "temp/wc_C.txt"]
expand의 첫 인자 내{filename}은 와일드카드가 아니고, 매개변수 리스트의 각 항목이 배정되는 위치이다.expand인자에서 와일드카드를 쓰려면 이중 중괄호{{ }}를 사용해야 한다.
Snakefile 은 기본적으로 파이썬 파일이기에, 다음과 같이 상수를 정의해 이용할 수 있다.
FILENAMES = ['A', 'B', 'C']
rule concat:
"""개별 단어 수 파일을 병합."""
input:
expand("temp/wc_{filename}.txt", filename=FILENAMES)
output:
"temp/wc_all.csv"
script:
"concat.py"
이 규칙에서는 세 파일을 결합하기 위해 concat.py 이라는 파이썬 파일을 실행해 temp/wc_all.csv 로 출력하는데, 코드는 다음과 같다.
import re
# wc -w 결과 파싱용 정규식
PTRN = re.compile(r'\s*(\d+)\s[^\s]+([^\s\/]+.txt)')
# 출력용 csv 파일 오픈
with open(snakemake.output[0], 'wt') as f:
f.write('fname, count\n')
# snakefile에 명시된 모든 입력 파일에 대해서
for fn in snakemake.input:
# 파싱하고 출력
line = open(fn, 'rt').read()
cnt, fn = PTRN.search(line).groups()
f.write('{}, {}\n'.format(fn, cnt))
각 입력 파일을 정규식으로 파싱한 후, .csv 형식에 맞게 출력 파일에 저장하고 있다.
Snakemake 를 통해 실행되는 파이썬 스크립트에는 snakemake 라는 전역 객체가 기본적으로 생성된다. 여기에는 Snakemake 의 실행 정보가 들어있다. 위 스크립트에서는 Snakefile 의 concat 규칙에서 정해진 입력 파일들을 얻어 오기 위해 snakemake.input 을, 출력 파일을 얻어 오기 위해서 snakemake.output 을 이용하고 있다. Snakefile 의 input 이나 output 에는 하나 이상의 파일이 올 수 있기에, 리스트로 취급한다.
위의 예처럼, 가급적 Snakemake 에서 제공하는 정보를 최대한 활용하는 것이 코드의 재활용성을 높여주는 좋은 습관이다.
이제 다음처럼 실행하면:
$ snakemake temp/wc_all.csv -j
data 디렉토리의 A.txt, B.txt, C.txt 세 파일의 단어 수가 하나의 temp/wc_all.csv 파일에 저장된다. 확인해보자.
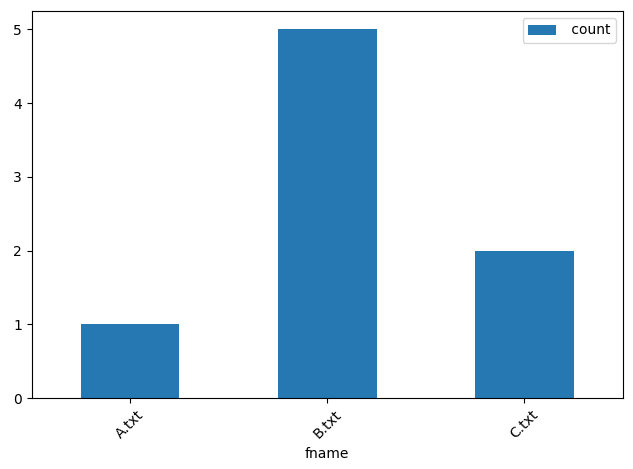
$ cat temp/wc_all.csv
fname, count
A.txt, 1
B.txt, 5
C.txt, 2
그래프 그리기
이제 위에서 생성된 temp/wc_all.csv 를 읽어 그래프를 그리는 마지막 규칙을 살펴보자. Snakefile 에 아래와 같은 규칙을 추가한다.
rule plot:
"""그래프 그리기."""
input:
"temp/wc_all.csv"
output:
"temp/wc_all.png"
script:
"plot.py"
스크립트파일 plot.py 는 temp/wc_all.csv 파일을 읽어 막대 그래프를 그리고 temp/wc_all.png 에 저장하는데, 코드는 아래와 같다.
import pandas as pd
df = pd.read_csv(snakemake.input[0], index_col='fname')
plot = df.plot(kind='bar', rot=45)
fig = plot.get_figure()
fig.tight_layout()
fig.savefig(snakemake.output[0])
실행하면 멋진(?) 그래프가 만들어진다.
$ snakemake temp/wc_all.png -j
만약 그래프 생성시
Unable to init server: Could not connect ... assertion 'GDK_IS_DISPLAY (display)' failed에러가 발생하면 아래 코드를plot.py상단에 추가한다.import matplotlib matplotlib.use('Agg')
기본 규칙 만들기
Snakemake 를 호출할 때마다 매번 타겟 파일을 지정하는 것은 번거롭다. Snakemake 는 타겟 파일이 없으면 첫 번째 규칙을 실행하는데, 다음의 규칙을 Snakefile 의 첫 규칙으로 추가해 기본 규칙 으로 동작하게 한다.
rule all:
"""기본 규칙."""
input: "temp/wc_all.png"
이 규칙에는 입력만 있는데, 그것을 생성하는 다른 규칙을 찾도록 하는 역할이기 때문이다. 이제 아래처럼 타겟 파일 없이 실행하면,
$ snakemake -j
자동으로 all 규칙이 선택되고, 그것의 입력인 temp/wc_all.png 를 생성하는 plot 규칙이 실행된다.
지금까지 작업한 전체 Snakefile 의 내용은 아래와 같다.
FILENAMES = ['A', 'B', 'C']
rule all:
"""기본 규칙."""
input:
"temp/wc_all.png"
rule count:
"""파일내 단어 수 세기."""
input:
"data/{filename}.txt"
output:
"temp/wc_{filename}.txt"
shell:
"wc -w {input} > {output}"
rule concat:
"""개별 단어 수 파일을 병합."""
input:
expand('temp/wc_{filename}.txt', filename=FILENAMES)
output:
"temp/wc_all.csv"
script:
"concat.py"
rule plot:
"""그래프 그리기."""
input:
"temp/wc_all.csv"
output:
"temp/wc_all.png"
script:
"plot.py"
DAG 시각화하기
위 워크플로우 파일의 DAG 를 다음처럼 시각화할 수 있다.
$ snakemake -j --dag | dot -Tpng -o dag.png
위에서
dot명령이 필요한데, 이것은 다음과 같이 설치 가능하다.sudo apt-get update sudo apt-get install graphviz
dag.png 를 열어보면 다음과 같다.
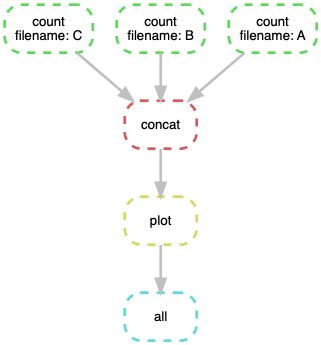
DAG 동작 돌아보기
지금까지 우리는 단계별로 진행을 했기에 중간 결과물들이 생성되어 있었지만, 처음부터 빌드하는 경우 다음과 같이 진행된다.
- 명시적 타겟 파일 없이 Snakemake 실행
- 첫 규칙인
all에서temp/wc_all.png파일을 입력으로 요구 - 그것을 출력하는
plot규칙에서temp/wc_all.csv파일을 입력으로 요구 - 그것을 출력하는
concat규칙에서temp/wc_A.txt,temp/wc_B.txt,temp/wc_C.txt파일을 입력으로 요구 - 그것들을 출력하는
count규칙에서 입력인data/A.txt,data/B.txt,data/C.txt의 단어를 세고 결과를 각각temp/wc_A.txt,temp/wc_B.txt,temp/wc_C.txt에 저장 - 입력이 해결된
concat규칙에서temp/wc_all.csv파일을 생성 - 입력이 해결된
plot규칙에서temp/wc_all.png파일을 생성
이러한 DAG 의존 관계를 통한 워크플로우는 순차적으로 작업을 하는 것이 아니라, 요청된 타겟 파일의 의존성을 쫓아 재귀적으로 진행되기에 일종의 On-Demand 또는 Lazy Evaluation 방식으로 볼 수 있겠다. 이 방식은 필요 없는 작업이 적고, 앞 규칙의 결과 파일을 이용하기에 여러 번 재시도해도 최단 시간에 결과를 확인할 수 있다.
예를 들어 앞의 plot 규칙에서, 좀 더 미려한 그래프를 그리기 위해서 코드를 여러 번 수정해야 하는 경우, (Jupyter 노트북처럼 인터랙티브한 환경이 아니라면) 매번 Python 을 실행하여 결과를 확인해야 한다. 이 과정에서 앞에서 실행했던 count 나 concat 이 불필요하게 실행될 수 있다. 물론, 코드를 수정하여 각 단계에서 중간 결과를 저장하고, 중간 결과가 있는 경우 그것을 이용하도록 할 수도 있겠으나, 아무래도 번거롭고 코드가 지저분해지게 된다. Snakemake 를 사용하면 의존 규칙의 결과인 temp/wc_all.csv 가 있으면 그래프 그리는 코드만 실행되기에 효율적이다.
심화
이제 Snakemake 에 대한 깊이 있는 활용 법을 알아보자.
두 가지 의존성
지금까지 막연하게 의존성을 이야기했으나, 사실 Snakemake 에서 다루는 의존성은 크게 두 가지로 나누어 설명할 수 있다. 그것은 데이터 의존성 과 워크플로우 의존성 이다 (이 두 용어는 필자가 설명의 편의를 위해 도입한 것이다) .
데이터 의존성
데이터 의존성은 타겟 파일 생성 후 입력 파일 (데이터) 이 변경되었다면 빌드가 무효화되는 것을 말한다. 변경 여부는 파일의 수정 시간을 기준으로 한다. Snakemake 는 데이터가 변하면 체크하여 자동으로 필요한 빌드를 다시 해준다. 예를 들어 아래와 같이 temp/A.txt 파일의 내용을 바꾸고,
Hello everybody.
Snakemake 를 다시 호출하면,
$ snakemake -j
변경된 data/A.txt 에 대해서만 count 규칙이 수행되고 data/B.txt 와 data/C.txt 에 대해서는 수행되지 않는다. 그후 갱신된 출력인 temp/wc_A.txt 에 의존하는 temp/wc_all.csv 와 그것에 의존하는 temp/wc_all.png 순으로 빌드가 진행되는 것을 볼 수 있다. 결과물인 그래프를 보면 A.txt 항목의 단어 수가 2 로 바뀐 것을 확인할 수 있다.
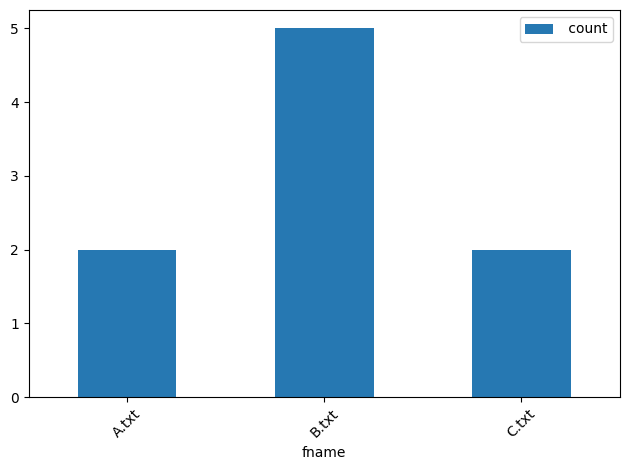
이렇게 의존성 체크를 통해 재작업이 필요한 부분만 다시 수행할 수 있는 것이 Snakemake 가 유용한 이유중 하나이다.
워크플로우 의존성
워크플로우 의존성은 Snakefile 에 기술된 규칙이나, 그것을 처리하는 코드에 변경이 있어 빌드가 무효화되는 것을 말한다. 예를 들어 다음처럼 concat.py 파일에 주석 DELETEME! 을 추가하고,
import re
# wc -w 결과 파싱용 정규식
PTRN = re.compile(r'\s*(\d+)\s[^\s]+([^\s\/]+.txt)')
# DELETEME!
# 출력용 csv 파일 오픈
with open(snakemake.output[0], 'wt') as f:
f.write('fname, count\n')
# snakefile에 명시된 모든 입력 파일에 대해서
for fn in snakemake.input:
# 파싱하고 출력
line = open(fn, 'rt').read()
cnt, fn = PTRN.search(line).groups()
f.write('{}, {}\n'.format(fn, cnt))
Snakemake 를 다시 실행해보면 다음 처럼 코드가 변경되었기에 다시 실행한다고 나온다.
[Fri Feb 16 15:51:28 2024]
rule concat:
input: temp/wc_A.txt, temp/wc_B.txt, temp/wc_C.txt
output: temp/wc_all.csv
jobid: 2
reason: Code has changed since last execution
resources: tmpdir=/tmp
[Fri Feb 16 15:51:28 2024]
Finished job 2.
Snakemake 는 .snakemake 디렉토리 아래 과거 결과물을 생성하는데 사용된 코드의 체크섬을 가지고 있다. 그것을 현재 코드의 체크섬과 비교하여 코드 변경 여부를 판단 후 재실행한 것이다.
규칙의 내 입력 및 매개변수 변화에 대해서도 같은 식으로 동작할 것이다.
사전형으로 입출력 기술하기
많은 입출력 파일을 사용하는 경우 리스트 형식으로 나열하는 것은 혼란스러울 수 있다. 이때는 키 = 값 형태로 입출력을 명시하면 편리하다.
rule RULE_NAME:
input:
foo_in="data/foo.txt"
boo_in="data/boo.txt"
output:
foo_out="temp/wc_foo.txt"
boo_out="temp/wc_boo.txt"
쉘 명령에서는 {input.foo_in} 또는 {output.foo_out} 처럼, 스크립트에서는 snakemake.input.foo_in 또는 snakemake.output.foo_out 처럼 입출력을 참조하면 된다.
규칙에 매개변수 이용하기
규칙에서 입출력 외에 실행 파트로 전달하고 싶은 값이 있을 때 params 키워드를 이용할 수 있다. input 처럼 하나 이상의 값이나, 코드 또는 함수가 올 수 있다. 아래는 와일드카드를 매개변수에 사용하는 예이다.
rule count:
"""파일내 단어 수 세기."""
input:
"data/{filename}.txt"
params:
fname="{filename}"
output:
"temp/wc_{filename}.txt"
shell:
"wc -w {input} > {output} && echo 'Done {params.fname}'"
스크립트 코드에서는 snakemake.params.fname 처럼 규칙의 매개변수를 참조하면 된다. 바뀐 코드를 실행하면 단어 수를 센 다음, 처리된 파일명을 표준 출력에 표시한다.
Done A
다음처럼 lambda 함수를 이용할 수도 있다.
rule count:
"""파일내 단어 수 세기."""
input:
"data/{filename}.txt"
params:
pair=lambda wildcards, output: "{} - {}".format(wildcards, output)
output:
"temp/wc_{filename}.txt"
shell:
"wc -w {input} > {output} && echo 'Done {params.pair}'"
결과는 다음과 같다.
Done A - temp/wc_A.txt
코드에서 와일드카드 참조하기
실행할 쉘 스크립트나 파이썬 코드에서 출력에서 얻은 와일드 카드를 매개변수 없이 직접 이용할 수도 있다.
다음과 같이 count 규칙을 수정후,
rule count:
"""파일내 단어 수 세기."""
input:
"data/{filename}.txt"
output:
"temp/wc_{filename}.txt"
shell:
"wc -w {input} > {output} && echo 'Done {wildcards.filename}'"
Snakemake 를 실행하면
snakemake -j
다음처럼 와일드카드 {filename} 에 해당하는 값이 출력되는 것을 확인할 수 있다.
# 동시 수행으로 출력 순서는 임의
Done B
Done C
Done A
특정 디렉토리내 모든 파일을 입력으로 하기
앞의 예에서는 어떤 입력 파일들이 있는지 알고 있다고 전제했다. 세 파일명 A, B, C 가 그것이었다. 만약 확장자는 같지만 임의 파일명으로 여러 파일이 있다면 어떻게 해야 할까? 파이썬의 glob 모듈은 패턴에 매칭되는 파일들을 찾아주는데, Snakemake 가 제공하는 glob_wildcards 도 비슷한 역할을 한다.
세 파일의 경우 아래의 코드를,
FILENAMES = ['A', 'B', 'C']
다음처럼 수정해도 같다.
FILENAMES, = glob_wildcards('temp/wc_{filename}.txt')
이렇게 하면 파일이 추가될 때도 워크플로우 파일 수정 없이 대응할 수 있는 장점이 있다.
객체가 아닌 파일명 리스트를 얻기 위해
FILENAMES다음에,가 있음에 주의하자.
와일드카드에 정규식으로 제약걸기
특정 타겟을 빌드할 때 타겟 이름을 요소별 와일드카드에 배정하고 싶을 때가 있다. 설명을 위해, 앞의 예제를 다음과 같이 바꾸어 생각해보자:
- 일별 파일들이
data/YYYYMMDD날짜형식 디렉토리 아래에 저장되고, - 일별 단어 수 그래프를
temp/YYYYMMDD/wc_all.png타겟으로 저장해야 한다.
예를 들어 다음과 같이 데이터가 있다면,
data/
20200401/
A.txt
B.txt
C.txt
20200402/
A.txt
B.txt
C.txt
20200403/
A.txt
B.txt
C.txt
특정 날짜, 예를 들어 2020년 4월 3일의 결과물을 snakemake temp/20200403/wc_all.png -j 명령으로 얻으려면, Snakefile 의 입력은 year, month, day 의 세 가지 와일드카드를 이용해 다음처럼 기술될 수 있을 것이다.
FILENAMES = ['A', 'B', 'C']
rule count:
input:
"data/{year}{month}{day}/{filename}.txt"
output:
"temp/{year}{month}{day}/wc_{filename}.txt"
shell:
"wc -w {input} > {output}"
rule concat:
input:
expand('temp/{{year}}{{month}}{{day}}/wc_{filename}.txt', filename=FILENAMES)
output:
"temp/{year}{month}{day}/wc_all.csv"
script:
"main.py"
rule plot:
input:
"temp/{year}{month}{day}/wc_all.csv"
output:
"temp/{year}{month}{day}/wc_all.png"
script:
"main.py"
concat규칙의expand에서 와일드카드를 이용하기 위해, 앞에서 말한 이중 중괄호를 사용하였다.
그러나 위와 같이 하면, Snakemake 는 year 가 4 자리, month 가 2 자리, day 가 2 자리인 것을 모르기 때문에 잘못된 와일드카드 배정으로 인한 오류나 RecursionError 가 발생할 수도 있다. 따라서, 안전한 방법은 {{와일드카드_이름, 정규식}} 형식으로 와일드카드의 패턴을 제한하는 것이다. 즉, 다음과 같이 할 수 있다.
FILENAMES = ['A', 'B', 'C']
rule count:
input:
"data/{year}{month}{day}/{filename}.txt"
output:
"temp/{year,\d{4}}{month,\d{2}}{day,\d{2}}/wc_{filename}.txt"
shell:
"wc -w {input} > {output}"
rule concat:
input:
expand('temp/{{year}}{{month}}{{day}}/wc_{filename}.txt', filename=FILENAMES)
output:
"temp/{year,\d{4}}{month,\d{2}}{day,\d{2}}/wc_all.csv"
script:
"main.py"
rule plot:
input:
"temp/{year}{month}{day}/wc_all.csv"
output:
"temp/{year,\d{4}}{month,\d{2}}{day,\d{2}}/wc_all.png"
script:
"main.py"
각 규칙의 출력에서 정규식으로 와일드카드의 값을 제약하고 있다. 와일드카드의 패턴을 제한하는 것은 출력 즉, output 에서만 사용할 수 있는데, 출력 와일드카드가 정해지면 입력은 그것을 그대로 쓰기 때문이다.
S3 에서 파일 입출력
클라우드 스토리지 서비스인 AWS S3 를 입출력 대상으로 사용할 수 있다. 앞의 예제를 S3를 이용하도록 다음처럼 바꾸어 생각해보자.
- 단어 수를 셀 텍스트 파일은
s3://my-bucket/data/아래에 있음 - 중간 산출물은 로컬의
temp/디렉토리에 출력 - 최종 타겟은
s3://my-bucket/temp/wc_all.png로 저장
예제의
my-bucket은 실제 자신이 사용할 버킷 이름으로 교체해야 한다.
이를 위해 Snakemake 에서 제공하는 S3RemoteProvider 를 사용한다. S3 URL 에서 s3:// 부분을 생략하고 다음처럼 수정한다.
from snakemake.remote.S3 import RemoteProvider as S3RemoteProvider
S3 = S3RemoteProvider(keep_local=True)
FILENAMES = ['A', 'B', 'C']
rule all:
input:
S3.remote("my-bucket/temp/wc_all.png")
rule count:
input:
S3.remote("my-bucket/data/{filename}.txt")
output:
"temp/wc_{filename}.txt"
shell:
"wc -w {input} > {output}"
rule concat:
input:
expand('temp/wc_{filename}.txt', filename=FILENAMES)
output:
"temp/wc_all.csv"
script:
"concat.py"
rule plot:
input:
"temp/wc_all.csv"
output:
S3.remote("my-bucket/temp/wc_all.png")
script:
"plot.py"
워크플로우 파일의 구성은 거의 같으나, S3RemoteProvider 를 사용해 S3 에 입출력을 하고 있다. 다음과 같은 방식으로 동작한다.
- 입력 파일이 S3 에 있는 경우, 먼저 Snakemake 가 그것을 동명의 로컬 디렉토리에 내려받는다. 실행 파트는 그 로컬 파일을 이용한다.
- 출력 파일이 S3 에 있는 경우, 먼저 실행 파트가 동명의 로컬 디렉토리에 파일을 출력한 뒤 Snakemake 는 그 파일을 S3 로 업로드 한다.
S3 입출력을 위해 임시로 사용한 로컬 파일은 더 이상 의존하는 규칙이 없으면 Snakemake 에 의해 지워진다.
S3RemoteProvier생성시keep_local=True으로 하면 지워지지 않는다.
빌드를 수행해보면, 현재 디렉토리 아래 my-bucket 디렉토리가 만들어진 것을 발견할 수 있다. 그 내용은 다음과 같다.
$ ls my-bucket/data/
A.txt B.txt C.txt
$ ls my-bucket/temp/
wc_all.png
이렇게 로컬 디렉토리를 경유하는 방식은, 각 쉘 명령이나 스크립트가 별도의 API 없이 S3 를 이용할 수 있게 해주기에 편리하다. 다만 크기가 큰 파일의 경우 로컬 디스크의 용량에 주의할 필요가 있겠다.
로컬 파일과 마찬가지로, 참조하는 S3 의 파일이 갱신되면 빌드가 무효화 된다 (keep_local 여부에 무관) .
위 예는 AWS CLI 툴의 설치 및 설정이 된 것을 가정하고 있다. 만약 그렇지 않다면,
S3 = S3RemoteProvider(access_key_id="MYACCESSKEY", secret_access_key="MYSECRET")식으로 AWS 계정 정보를 넣어 주어야 한다.
다음처럼
S3RemoteProvider를 통해서도expand를 수행할 수 있다.
S3.remote(expand("my-backet/data/{filename}.txt", filename=FILENAMES))
Jupyter 노트북 실행하기
Snakemake 는 Jupyter 노트북으로 구현된 코드를 실행 파트로 사용할 수 있다. concat 규칙의 실행 파트를 Jupyter 노트북 파일로 바꾸어 보자. 아래처럼 script 를 notebook 키워드로 수정한다.
"""개별 단어 수 파일을 병합."""
rule concat:
input:
expand('temp/wc_{filename}.txt', filename=FILENAMES)
output:
"temp/wc_all.csv"
notebook:
"concat.ipynb"
파이썬 파일 대신 Jupyter 노트북 파일인 .ipynb 가 사용되었다. 이제 이 파일을 만들어주면 되는데, 문제점은 노트북에서 개발할 때는 snakemake 객체가 없다는 것이다. 개발할 때는 명시적으로 파일명을 이용하고, 빌드할 때는 snakemake 를 참조하도록 수정할 수 있겠으나, 아무래도 실수의 여지가 많다. 하나의 아이디어는 개발시에만 동작하는 snakemake 의 막업 (Mockup) 객체를 만드는 것이다.
# snakemake 막업 (Mockup) 만들기
class AttrDict(dict):
def __getattr__(self, attr):
return self[attr]
def __setattr__(self, attr, value):
self[attr] = value
# Snakemake 로 실행되지 않을 때만 막업 사용
if 'snakemake' not in globals():
snakemake = AttrDict()
# 노트북 개발시 코드 동작을 확인할 수 있는 적당한 입출력
snakemake.input = ['temp/wc_A.txt', 'temp/wc_B.txt']
snakemake.output = ['temp/wc_all.csv']
이것을 사용하면 concat 규칙을 위한 노트북 파일 concat.ipynb 는 아래와 같은 모습이 될 것이다.
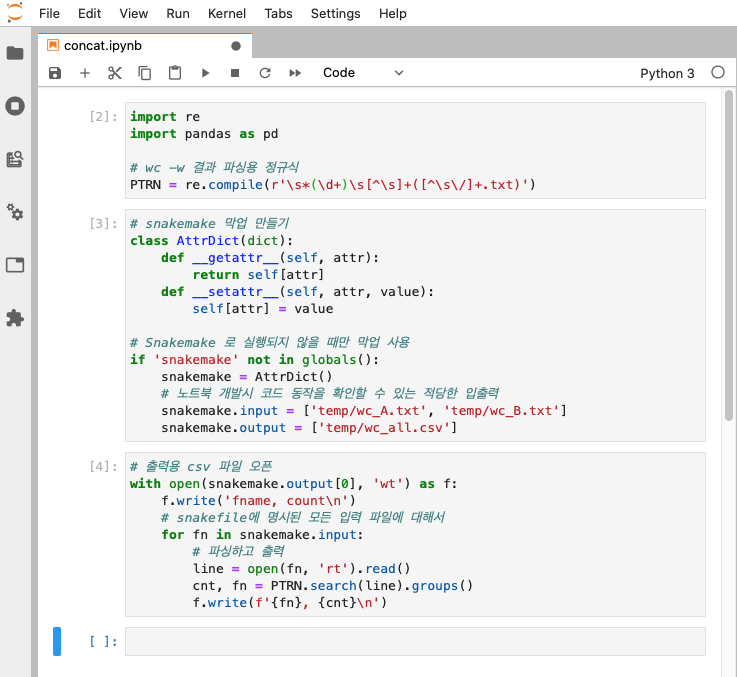
이제 노트북 개발시에는 막업의 정보를, Snakemake 를 통한 실행시에는 완전한 정보를 사용하게 된다.
Jupyter 노트북은 훌륭한 툴이지만, 하나의 노트북 파일에서 모든 것을 구현하려는 경향이 생기기 쉽고 코드 재활용성이 떨어지기에, 필자는 Snakemake 와 Jupyter 노트북을 함께 사용하는 것은 추천하지 않는다.
외부 워크플로우 파일 이용하기
하나의 Snakefile 에서 외부의 Snakefile 을 서브워크플로우 (Subworkflow) 로 이용하는 것이 가능하다. 이 기능은 빌드를 계층화하거나, 여러 사용자가 협업할 때 유용할 것이다.
앞의 예를 데이터 엔지니어 (u1) 와 데이터 분석가 (u2) 가 나누어 작업하는 경우로 바꾸어 생각해보자. 엔지니어는 데이터 파일을 읽어 단어 수 파일 (wc_all.csv) 을 만들고, 분석가가 이를 이용해 시각화 파일 (wc_all.png) 을 만드는 것으로 가정한다. git 등으로 코드 관리에 용이하게 다음처럼 디렉토리를 구성한다.
u1/
Snakefile
concat.py
data/
A.txt
B.txt
C.txt
temp/
u2/
Snakefile
plot.py
temp/
데이터 엔지니어 (u1) 의 Snakefile 은 다음과 같다.
FILENAMES = ['A', 'B', 'C']
rule u1all:
"""사용자 1 의 기본 규칙."""
input:
"temp/wc_all.csv"
rule count:
"""파일내 수 세기."""
input:
"data/{filename}.txt"
output:
"temp/wc_{filename}.txt"
shell:
"wc -w {input} > {output}"
rule concat:
"""개별 단어 수 파일을 병합."""
input:
expand('temp/wc_{filename}.txt', filename=FILENAMES)
output:
"temp/wc_all.csv"
script:
"concat.py"
앞에서 본 예와 크게 다르지 않으나, concat 규칙까지만 구현하고 기본 규칙의 이름을 u1all 로 한 것이 눈에 띄인다. 이는 다른 워크플로우 파일에 포함되어 사용할 때, 같은 이름의 규칙이 있으면 충돌이 일어나기 때문이다.
데이터 분석가 (u2) 의 Snakefile 은 아래와 같다.
subworkflow data:
workdir:
"../u1"
snakefile:
"../u1/Snakefile"
rule u2all:
"""사용자 2 의 기본 규칙."""
input:
"temp/wc_all.png"
rule plot:
"""그래프 그리기."""
input:
data("temp/wc_all.csv")
output:
"temp/wc_all.png"
script:
"plot.py"
데이터 엔지니어 (u1) 의 워크플로우를 subworkflow 를 이용해 서브워크플로우로 선언하고, 그것의 최종 타겟인 temp/wc_all.csv 를 입력으로 하여 plot 규칙을 정의하고 있다. 서브워크폴로우의 이름인 data 를 함수처럼 사용해, 포함된 워크플로우의 결과 파일을 참조하는 것에 주의하자.
기본 규칙은 include 된 파일의 것은 무시하고, 현재 워크플로우의 첫 규칙인
u2all이 선택된다.
데이터 분석가의 디렉토리에서 다음처럼 빌드를 수행하면,
$ cd u2/
$ snakemake -j
데이터 엔지니어의 빌드가 먼저 수행되고 작업 결과는 u1/temp 에 저장된다. 이어 데이터 분석가의 빌드가 수행되고 작업 결과는 u2/temp 에 저장된다. 이런 식으로 중복 작업없이 다른 사용자와 효과적인 협업이 가능할 것이다.
설정 파일 이용하기
어떤 상수가 빌드시 자주 변경되거나 하나 이상의 워크플로우 파일에서 공유될 수 있다면, 설정 파일에 기술하는 것이 바람직하다. 예를 들어 두 프로젝트 A, B 의 데이터 처리 (ETL) 를 위해 다음과 같은 디렉토리 구조로 각각의 워크플로우와 스크립트 파일을 만들어 사용하고 있다고 하자.
projects/
A/
Snakefile
etl.py
temp/
B/
Snakefile
etl.py
temp/
프로젝트 A 의 Snakefile
EC2TYPE = 'm5.large'
rule etl:
output:
"temp/result.csv"
params:
ec2type=EC2TYPE
script:
"etl.py"
프로젝트 B 의 Snakefile
EC2TYPE = 'm5.large'
rule etl:
output:
"temp/result.csv"
params:
ec2type=EC2TYPE
script:
"etl.py"
etl.py 에서는 매개변수로 전달된 ec2type 에 맞게 클라우드 인스턴스를 띄우고, 처리 작업을 진행하는 코드가 구현되어 있다고 하자.
먼저 보이는 것이 EC2TYPE 이 중복되고 있다는 것이다. 이것을 상위 디렉토리의 별도 설정 파일 (config.yaml) 에 다음처럼 기술한다. 워크플로우 파일에서 configfile 으로 선언하고, config 를 통해 참조할 수 있다.
projects/
config.yaml
A/
Snakefile
etl.py
temp/
B/
Snakefile
etl.py
temp/
설정 파일 config.yaml
ec2type: "m5.large"
프로젝트 A 의 Snakefile
configfile: "../config.yaml"
rule etl:
output:
"temp/result.csv"
params:
ec2type=config['ec2type']
script:
"etl.py"
프로젝트 B 의 Snakefile
configfile: "../config.yaml"
rule etl:
output:
"temp/result.csv"
params:
ec2type=config['ec2type']
script:
"etl.py"
이제 중복된 상수를 설정 파일을 통해 공유하게 되었다. 사실 설정 파일을 사용하면 스크립트 코드에서 매개변수를 통하지 않고, snakemake.config['ec2type'] 처럼 설정값을 바로 참조할 수 있기에 워크플로우 파일은 더 단순해진다.
프로젝트 A 의 Snakefile
configfile: "../config.yaml"
rule etl:
output:
"temp/result.csv"
script:
"etl.py"
프로젝트 B 의 Snakefile
configfile: "../config.yaml"
rule etl:
output:
"temp/result.csv"
script:
"etl.py"
어느날 데이터가 늘어 빌드시 큰 인스턴스를 사용해야 한다면, 다음과 같이 기본 설정값을 덮어쓸 수 있다.
$ snakemake -R etl -j --config ec2type=m5.2xlarge
Snakemake 는 설정값이 바뀌었어도 입력 파일이 바뀌지 않았으면 새로 빌드하지 않는다. 바뀐 설정값을 적용하기 위해
-R을 사용하여 명시적으로 빌드를 하게 하자.
스크립트 파일 합치기?
규칙마다 파이썬 파일을 하나씩 만들어 주는 것은 언듯 귀찮아 보인다. 매번 파일을 만드는 것도 일이지만, 작업을 위한 기본 코드 (Boilerplate) 가 많다면 더 힘들어지기 때문이다. snakemake.rule 속성에 현재 실행 규칙의 이름이 오는 것을 용해 여러 규칙의 코드를 하나의 파일에 합치는 것이 가능하기는 하다.
그러나 필자는 이것을 추천하지 않는다. 왜냐하면 여러 규칙에서 하나의 스크립트 파일을 공유하면 스크립트의 일부만 변경되어도 그것을 참조하는 모든 규칙이 무효화되어, Snakemake 의 빌드 효율성을 크게 저하하는 결과가 되기 때문이다.
반복적인 코드는 공용 모듈에 리팩토링하여 공유하는 식으로 하고, 가급적 규칙 당 하나의 스크립트 파일을 사용하는 것을 권장한다.
기타 팁들
쉘 명령어 여러 줄 사용하기
다음처럼 멀티라인 문자열로 한 줄 이상의 쉘 명령어를 사용할 수 있다.
shell:
"""
command 1
command 2
command 3
"""
더미 출력 파일 이용하기
Snakemake 는 모든 작업의 결과가 파일로 남는 것을 전제로 하고 있지만, 파일을 출력하지 않는 작업도 있을 것이다. 이런 경우 다음처럼 더미 출력 파일 을 생성하는 규칙을 만들어 사용할 수 있다.
rule wait:
input:
"temp/wc_all.png"
output:
"temp/wait_done"
shell:
"""
sleep 4
touch temp/wait_done
"""
더미 출력 파일은 보통 결과 파일 생성이 목적이 아니라 Snakemake 를 통해 특정 명령을 실행하고자 할 때 유용하다. 따라서 다음처럼 결과 파일의 존재 여부에 무관하게 실행되도록 -f 를 붙여서 실행하는 것이 일반적이다.
snakemake -f temp/wait_done -j
강제로 모든 규칙 실행하기
--forceall 줄여서 -F 옵션으로 유/무효 여부에 관계없이 모든 규칙을 실행할 수 있다.
$ snakemake -j -F
고정된 출력 파일의 입력을 함수에서 얻기
입력은 함수를 통해 동적으로 얻고, 그 결과를 처리하는 경우를 생각해보자.
아래는 10 분 단위로 입력 파일이 변하는 규칙이다.
from datetime import datetime
def get_input(wc, now):
# 입력 파일 리스트 만들고 반환
return [os.path.join('data', now.strftime('%H%M'))[:-1]]
rule timed_input:
"""시간에 따라 입력 파일을 변경."""
input:
lambda wc: get_input(wc, datetime.now())
output:
"temp/timed_input.txt"
shell:
"stat {input} > {output} && echo 'Done'"
이 규칙의 실행을 위해서는 data/ 디렉토리 아래에 10분 단위 숫자로된 더미 입력 파일이 있어야 한다. 예를 들어 현재 16 시 38 분이라면 다음과 같은 파일을 만들어 주자.
touch data/163
다음과 같이 실행하면,
snakemake temp/timed_input.txt -j
최초 한 번은 바로 빌드가 되지만, 다음 부터는 최대 10분 간격으로만 실행되는 것을 확인할 수 있다.
이렇게 Snakemake 는 함수를 활용한 동적 입력도 잘 처리한다.
체크포인트 이용하기
만약 어떤 규칙에서 이후 규칙에서 처리해야할 파일 목록이 담긴 출력 파일을 생성하고, 다른 규칙에서 그것을 입력으로 사용하되 병렬처리가 가능해야하는 경우는 어떻게 해야 할까? expand 를 이용해야 하겠지만, 처리해야할 파일 리스트가 확정되지 않는 상황이기에 쉽지 않다.
일반적인 snakemake 워크플로우에서는 모든 규칙과 파일이 사전에 정의되어 있어야 한다. 그러나 일부 워크플로우에서는 위 예처럼 중간 단계에서 생성된 파일이나 결과에 따라 후속 작업이 결정되는 경우가 있다. 이러한 경우 체크포인트 (checkpoint) 를 사용하여 동적으로 워크플로우를 정의할 수 있다.
checkpoint generate_file_list:
"""파일 목록 생성."""
output:
"file_list.txt"
shell:
"""
echo "file1.txt" > {output}
echo "file2.txt" >> {output}
"""
rule all:
"""파일 목록을 기반으로 후속 작업 병렬 실행."""
input:
expand("processed_{file}", file=checkpoint(generate_file_list).get().output)
rule process_file:
"""개별 파일 처리."""
output:
"processed_{file}"
run:
with open(output[0], "w") as out_f:
out_f.write(f"Processed {file}")
generate_file_list: 체크포인트 규칙으로 파일 목록을 생성하고file_list.txt파일에 저장한다.all: 최종 목표를 정의하는데,checkpoint(generate_file_list).get().output을 사용하여generate_file_list에서 생성된 파일 목록을 기반으로 후속 작업을 동적으로 실행되게 한다.process_file: 개별 파일에 대한 처리
이런 방식으로 구현하면 확정된 파일명이 없고, 선행 규칙의 결과로 입력이 얻어지는 상황에서도 의존관계 설정 및 병렬 처리를 구현할 수 있다.
빌드 결과물 모두 지우기
예제처럼 하나의 디렉토리에 중간 결과물과 최종 타겟이 모두 저장되는 경우는 디렉토리 자체를 지우면 되나, 산출물 디렉토리를 몇 개로 구분해서 사용하는 경우에는 번거로울 수 있다. 이때는 아래 명령으로 빌드시 생성된 모든 파일을 제거할 수 있다.
$ snakemake -j --delete-all-output
메모리 문제 대응
메모리를 많이 사용하는 규칙을 병렬로 실행하면, 시스템 메모리를 금방 소진해 프로세스가 kill 되기 쉽다. 이런 때는 규칙의 resources 파트에 규칙이 사용하는 대강의 메모리 정보를 기술하는 것이 도움이 된다. 먼저 메모리를 많이 사용하는 규칙에 대해, 다음처럼 하나의 타겟을 빌드하면서 필요 메모리를 확인한다:
$ /usr/bin/time -v snakemake -f temp/20210307/big_data.parquet -j
위 예는 하루치 big_data.parquet 파일을 빌드하는 것인데, 전체 날자를 빌드하기 위한 더미 파일은 all_big_data 라고 하겠다. 출력에서 Maximum resident set size (kbytes) 을 찾는다. 위 명령에서는 아래와 같았다:
Maximum resident set size (kbytes): 11768224
위의 경우 하루치 데이터에 대해 최대 11.5 GB 정도의 메모리를 사용하고 있다. Snakefile 의 규칙에서 다음과 같이 MB 단위로 메모리 사용량을 지정한다.
rule big_data:
..
resources:
mem_mb=11492 # MB 단위
이런 식으로 메모리를 많이 사용하는 규칙에 대해 메모리 사용량을 기입한다. 다음은 시스템의 가용 (free) 메모리를 확인한다:
$ free
total used free shared buff/cache available
Mem: 32472084 320252 27017500 9348 5134332 31721596
Swap: 0 0 0
여기서는 free 메모리가 가 27,017,500 (KB) 이다. 이것을 인자로 하여 Snakemake 를 실행한다. 메모리 사용량이 지정되지 않은 다른 규칙도 있다면, 실제 가용 메모리 보다는 좀 줄여서 MB 단위로 기입한다.
$ snakemake -f temp/all_big_data -j --resources mem_mb=24000
이렇게 하면 Snakemake 가 최대 가용 메모리가 넘지 않도록 작업을 실행하기에, 메모리 에러가 발생하지 않을 것이다 (이 예에서는 아마도 규칙 두 개가 정도가 동시에 실행될 것이다).
만약 작업 중 메모리 에러나 프로세스 kill 이 발생하면, -–resources mem_mb 인자의 값을 더 줄여서 다시 시도해보자.
마무리
Snakemake 에는 여기에 설명하지 않은 많은 기능이 있다. 공식 문서 및 검색을 통해 Snakemake 의 강력한 기능을 파악하고 활용하도록 하자.